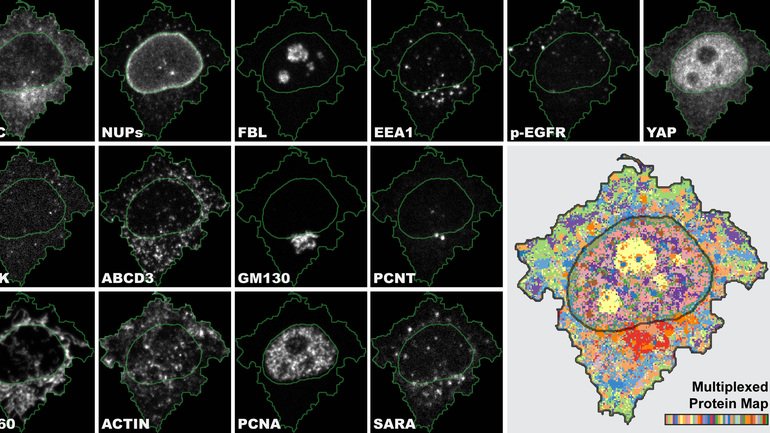
Forscher der Universität Zürich haben eine neuartige Methode zur Untersuchung von Zellen und ihren Bestandteilen entwickelt, die sogenannte „Iterative Indirect Immunofluorescence Imaging“(4i). Diese Innovation verfeinert die in der Biomedizin übliche Analysemethode der Immunfluoreszenz um ein Vielfaches und ermöglicht, innerhalb einer Probe enorme Datenvolumen zu gewinnen. Mit 4i lässt sich die räumliche Verteilung von mindestens 40 Proteinen und deren Veränderungen in derselben Zelle gleichzeitig für Hunderttausende von Zellen beobachten. Und zwar auf verschiedenen Ebenen – vom Zellgewebe bis zu den Zellorganellen.
Zehnmal mehr Proteine gleichzeitig visualisieren
„4i ist das erste bildgebende Verfahren, das eine enorm vielschichtige Ansicht von biologischen Proben ermöglicht. Zum ersten Mal können wir multiplexierte Informationen, die auf Gewebe-, Zell- und Organell-Ebene gewonnen werden, in ein und demselben Experiment miteinander verbinden“, sagt Gabriele Gut, Hauptautor und Postdoktorand am Institute of Molecular Life Sciences der UZH.
Die Technik der Immunfluoreszenz verwendet Antikörper, um Proteine von biologischen Proben sichtbar zu machen und zu lokalisieren. Während mit der herkömmlichen Immunfluoreszenz in der Regel gleichzeitig drei Eiweiße markiert werden, ist 4i mit handelsüblichen Antikörpern und konventionellen Fluoreszenzmikroskopen in der Lage, durch schrittweise Applizierung und Wieder-Entfernung von Antikörpern zehnmal mehr Proteine in derselben Probe gleichzeitig zu visualisieren.
Karte gibt Übersicht über die Zelllandschaft
Sind die Daten erfasst, muss die riesige Menge auch analysiert werden können – die nächste Hürde für die Forschenden. „Wir generierten Bilder mit subzellulärer Auflösung für Tausende von Zellen über 40 Kanäle für mehr als 10 verschiedene Bedingungen. Das menschliche Auge und das Gehirn können die mithilfe von 4i gesammelte biologische Komplexität nicht mehr verarbeiten“, sagt Gabriele Gut.
Um die 4i-Daten vollständig zu nutzen, entwickelte er daher ein neues Analyse- und Visualisierungsprogramm namens „Multiplexed Protein Maps“. Dieses ermittelt das Multiplex-Fluoreszenzsignal für Millionen von Pixeln und erzeugt damit eine abstrakte, aber repräsentative Karte der Verteilung dutzender verschiedener Proteinen in der Zelle. Die Forscher erstellten damit eine systematische Übersicht über die Zelllandschaft: Sie konnten die räumliche intrazelluläre Organisation der meisten Säugetierorganellen während der verschiedenen Stadien des Zellzyklus‘ sowie in verschiedenen Mikroumgebungen darstellen.
Personalisierte Medizin und Tumortherapie
Die Anwendungen für 4i und Multiplex-Proteinkarten reichen von der Grundlagenforschung bis zur personalisierten Medizin. 4i kann auch ermitteln, wie sich pharmakologische Substanzen auf die Organisation und Physiologie der Zellen auswirken. Sie wird derzeit in einer Forschungszusammenarbeit mit Klinikern und einem Pharmaunternehmen eingesetzt, um die Behandlung von Krebspatienten zu verbessern. Dabei werden Tumorzellen von Patienten untersucht, die mit verschiedenen Krebsmedikamenten behandelt wurden.
http://science.sciencemag.org/content/361/6401/eaar7042
www.media.uzh.ch/de/medienmitteilungen/2018/Verfeinerte_Analyse.html