Ein paar wenige Zellen, die anders sind als der Rest, können große Auswirkungen haben. So können etwa einzelne Krebszellen einer Chemotherapie gegenüber unempfindlich sein und einen Rückfall bei eigentlich als geheilt geltenden Patienten verursachen. Molekulare Analysen einzelner Zellen sind daher notwendig, um die Rolle gemischter Zellansammlungen bei der Entstehung von Krankheiten besser zu verstehen und um effektive Therapien für eine personalisierte Medizin zu entwickeln.
Doch individuelle Zellen in einer Masse anderer Zellen zu identifizieren, stellt die Diagnostik vor enorme Herausforderungen. Die Zellen müssen sortiert, festgehalten, in ein anderes Gefäß mit extrem geringem Volumen (1 μl) überführt und anschließend molekular analysiert werden. Konventionelle Methoden sind meist zeitaufwändig und komplex, unzuverlässig und ineffizient, können die Lebensfähigkeit der Zellen beeinträchtigen, benötigen große Probenvolumina, bergen ein hohes Kontaminationsrisiko und/oder erfordern teure Geräte.
Mikrofluidtechnik umgeht die Probleme
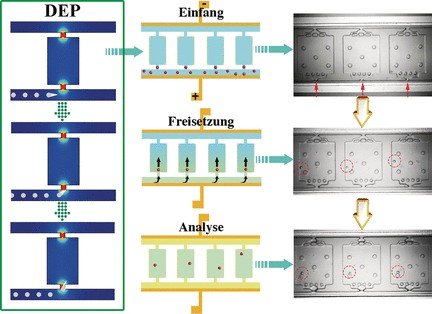
Die Wissenschaftler von der University of Washington in Seattle, USA, der Iowa State University in Ames, USA, und dem Fred Hutchinson Cancer Research Center in Seattle, USA, setzen auf Mikrofluidtechnik, um diese Probleme zu umgehen. Alle notwendigen Schritte erfolgen zuverlässig auf einem speziell entwickelten Mikrochip unter Verwendung minimaler Reagenzienmengen und ohne die Zellen markieren zu müssen. Anders als bei herkömmlichen Mikrofluidchips werden weder komplexe Fabrikationstechniken noch Bauteile wie Ventile oder Mischer benötigt.
Der sogenannte Self-Digitization-Dielectrophoretic-Chip (SD-DEP-Chip) ist nur etwa so groß wie eine Münze und trägt zwei parallele Mikrokanäle (50 μm tief × 35 μm breit × 3,2 cm lang), die über eine Vielzahl winziger Kämmerchen verbunden sind. Die Öffnungen der Mikrokammern sind nur 15 μm schmal. Je eine dünne Elektrode erstreckt sich über die Länge der Kanäle. Kanälchen und Kämmerchen werden mit einem Puffer gefüllt, eine elektrische Wechselspannung angelegt und die Probe in einen der Mikrokanäle gespült. Als Beispiel verwendete das Team um Robbyn K. Anand und Daniel T. Chiu Leukämie-Zellen.
Zellen mit elektrischem Feld fangen
An den schmalen Eingängen zu den Kammern treten lokale Maxima des elektrischen Felds auf. Zellen, die hinein geraten, werden „gefangen“. Da die Öffnungen etwa so groß sind, wie die Zellen, wird immer nur eine einzelne Zelle pro Kammereingang festgehalten. Durch Abstellen des Wechselstroms und Erhöhen der Fließgeschwindigkeit durch Injektion der für die spätere Analytik benötigten Reagenzien werden die Zellen in die Kammern gespült. Anschließend eingeleitetes Öl verschließt die Kammern. Die Zellen werden aufgelöst, die freigesetzten Nukleinsäuren vervielfältigt und können zum Beispiel über ein Marker-Gen als Leukämie-Zellen identifiziert werden.
In folgenden Studien wollen die Forscher den Chip einsetzen, um die Verteilung genetischer Mutationen in Leukämie-Zellen zu bestimmen, die mit Resistenzen in Zusammenhang stehen und somit für Rückfälle verantwortlich sein könnten.
https://doi.org/10.1002/ange.201807314
http://depts.washington.edu/chem/people/faculty/chiu.html